TD4 : Electrophorèse
Expérience 2 : Gel 2D d’extraits
protéiques de biofilm de Streptococcus mutans.
|
But de l’expérience :
Afin de déterminer les changements
protéomiques dans les biofilms formés par
Streptococcus mutans, consécutifs à un choc
acide. S. mutans est l’une des principales causes de
caries dentaires. Un biofilm de S. mutans se forme
notamment sur la plaque dentaire. Plus il y a de bactéries
S. mutans et plus le risque de caries est important.
Découvrir les changements protéiques qui protègent
S. mutans durant l’acidification (qu’elle
génère elle-même) permettrait peut-être
de cibler son moyen de protection et donc, d’éliminer
cette bactérie.
|
Carie.
(lien carie)
|
|
Question : Pourquoi effectuer
un marquage radioactif alors qu’une coloration peut être
effectuée ?
|
Avant-propos: Consignes
Cette expérience virtuelle se déroule de manière
similaire à la première :
Cette expérience virtuelle est inspirée de
véritables expériences. Cependant, afin de rendre le TD
plus intéressant et de favoriser les questions, des
adaptations ont été effectuées (dont certaines
sont peu probables).
Article ayant inspiré cette
expérience : Welin
et al. Effect of acid shock on protein expression by biofilm cells
of S. mutans. FEMS microbiology letters. 2003
Introduction :
Convention : 1 kDa (kilo Dalton) = 1'000 Da (Dalton, unité
de mesure de masse moléculaire). pI = point Isoélectrique,
il correspond au pH pour lequel une protéine est globalement «
neutre » (autant de charges + que de charges –).
Streptococcus mutans :
Streptococcus mutans est une bactérie de type «
coque à Gram + », présente notamment dans la
région buccale et en particulier sur la plaque dentaire, où
elle forme des biofilms et acidifie son environnement (ce qui détruit
l’émail des dents et provoque les caries). S. mutans
est anaérobe et aérotolérante (quelle
différence ?), elle se présente en général
en petite chaîne de coques. En tant qu’anaérobe,
elle produit son énergie à partir de la dégradation
du glucose en acide lactique (voyez-vous
un lien avec la formation de caries ?)
Le génome de S. mutans représente 2Mb,
entièrement séquencé en 2002 (lien
; lien),
et contient environ 2000 gènes.
Le temps de génération (doublement) de S. mutans
est de 7h, dans les conditions utilisées pour cette expérience
(et donc très loin des 20min. de E. coli, utilisée
dans le
TD2).
Plus d’informations sur les streptocoques :
http://membres.lycos.fr/neb5000/BacteriologieI/Groupes%20Bacteriens/Coques%20Gram-positifs.htm
Plus d’informations sur la formation de caries :
http://www.caobisco.com/french/pdf/Caries.pdf
Le Protéome :
Il y a deux approches majeures de l’étude des
organismes, l’une génétique (à partir des
gènes) et l’autre protéique (à partir des
protéines). L’ensemble des gènes d’un
organisme représente son génome. De manière
similaire, l’ensemble des protéines contenues dans une
cellule à un moment donné, représente son
protéome. Il y a donc plusieurs protéomes pour chaque
organisme, car celui-ci varie en fonction de l’activité
de la cellule et de son environnement. On trouve des protéomes
sur internet, notamment sous forme de gels 2D colorés (lien).
En résumé, le génome vous donnera une
information sur les possibilités d’une cellule, alors
que son protéome vous donnera sa composition exacte à
un moment très précis.
Le marquage radioactif des protéines :
Il existe plusieurs moyens de marquer des protéines.
L’incorporation d’éléments radioactifs
durant la synthèse des protéines par l’organisme
étudié, permet de marquer spécifiquement les
protéines nouvellement synthétisées. En effet,
si vous utilisez une coloration non spécifique, vous
détecterez toutes les protéines, «anciennes et
nouvelles». Dans certains cas, il peut être intéressant
de ne marquer que les «nouvelles» protéines. Il
existe deux isotopes radioactifs couramment utilisés pour
marquer les protéines : 14C et 35S. En
général, on fabrique des acides aminés contenant
l’un des deux isotopes, que l’on place ensuite dans le
milieu de cultures des bactéries. Ces dernières les
incorporent alors dans leurs protéines nouvellement
synthétisées. Un autre avantage de l’utilisation
de la radioactivité c’est qu’il n’est pas
nécessaire d’utiliser une méthode de coloration
pour « visualiser les protéines marquées ».
En effet, la fission des isotopes radioactifs émet des
rayonnements suffisants à impressionner un film
photographique. Si cela ne vous semble pas clair, un schéma
explicatif sera donné durant la synthèse de
l'expérience.
Question : Voyez-vous une différence
stratégique entre l’utilisation du 14C et
celle du 35S ? Sachant qu’il n’y a pas de
grandes différences d’intensité de radiations
produites par ces deux isotopes.
Expérience 2
Etape 1 : Marquage des protéines
La première étape consiste à marquer les
protéines avec l’isotope radioactif. Pour cela, nous
utilisons une méthode dite de « pulse », où
des nutriments radioactifs sont donnés aux cellules pendant un
certain temps.
Nous effectuons 2 cultures de S. mutans, la première
favorisant une croissance dite planctonique alors que la seconde
favorise la création de biofilms. Pour chacun de ces systèmes,
nous effectuons une culture à pH 7.5 et une autre à
5.5. Vous disposez donc de 4 cultures différentes. (pourquoi
en faire autant ?).
Les cellules sont donc incubées en présence d’acides
aminés marqués radioactivement (quelles
mesures de protection devez-vous prendre ?) par du 14C
pendant 2 heures (c’est le « pulse »). Après
ce marquage, les cellules sont récupérées et
lysées par ultrasonication (les cellules sont soumises à
un traitement aux ultrasons qui cassent leur paroi, et un détergent
dissout leur membrane). Les protéines sont alors récupérées
pour la suite de l’expérience.
Après la lyse, avez-vous des protéines non-marquées
radioactivement, et si oui, devez-vous les éliminer avant de
poursuivre votre expérience ? (choisissez la bonne
illustration)
|
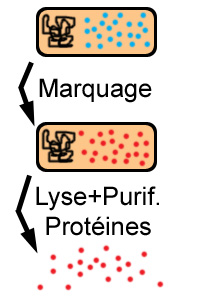
Non, toutes les protéines récupérées
sont marquées.
|
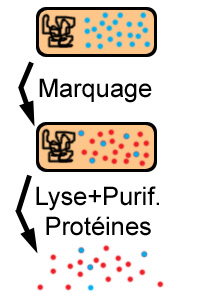
Oui, il y a des protéines non-marquées,
mais il n’est pas nécessaire de les enlever pour la
suite de l’expérience.
|
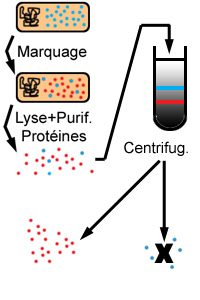
Oui, il y a des protéines non-marquées.
Il faut séparer les protéines marquées des
protéines non-marquées en centrifugeant l’extrait
sur un gradient de Chlorure de Cesium (CsCl ; les protéines
marquées par le 14C sont plus denses que celles
contenant du 12C, il est donc facile de les séparer).
|
B. Emery. 24.11.05