TD4 : Electrophorèse
Expérience 1 : Construction d'une banque
d’ADN génomique de Candida albicans.
Avant-propos: Consignes :
- Besoin de révisions ? C'est par ici : Introduction
- Vous disposez de 25 minutes pour réaliser l’expérience.
Ne perdez pas trop de temps.
- Lisez attentivement la description de l’expérience.
Lorsqu’on vous demandera de faire un choix, réfléchissez
et cliquez sur la réponse qui vous paraît la plus
logique.
- Même si vous avez répondu correctement, pensez à lire les autres
choix.
- Les réponses aux questions en italique figurent
généralement sur une autre page html (cliquez sur la
question). Ne vous attardez pas sur ces questions.
- Les questions en rouge seront discutées durant la
synthèse de l’expérience.
- Si vous ne terminez pas l'expérience dans le temps imparti, vous pourrez la refaire plus tard.
L'adresse web de l'expérience est en lien sur Dokeos.
- Vous pourrez toujours m'adresser vos remarques par email : Bertrand.Emery@romandie.com.
Cette expérience virtuelle est inspirée par de
véritables expériences, cependant, afin de rendre le TD
plus intéressant et favoriser les questions, des adaptations
ont été effectuées (dont certaines sont peu
probables).
Articles ayant inspiré cette
expérience : Richard
et al. Candida albicans Biofilm-defective mutants. Euk. Cell,
2005. Davis
et al. C. albicans Mds3p, a conserved regulator of pH responses
and virulence identified through insertional mutagenesis.
Genetics 2002
Introduction :
Convention : 1Mb {méga (paires de) bases} = 1'000 kb
{kilo (paire de) bases} = 1'000'000 pb {paire de bases}. Il s’agit
ici d’un moyen de quantifier la taille de molécules
d’ADN, en nucléotides appariés. Chaque acide
aminé (aa) est codé (sur l’ADN/ARN) par un groupe
de 3 nucléotides (=codon). Un ORF (=cadre de lecture ouvert ;
partie du gène codant pour la protéine) mesurant ~1kb
code donc pour une protéine de ~332 aa (pourquoi
pas 333 ?).
Candida albicans :
Candida albicans est un champignon unicellulaire (eucaryote
inférieur) diploïde, croissant sous forme unicellulaire
et d’hyphe (filaments de cellules reliées entre-elles).
Le biofilm formé par C. albicans est composé de
cellules unicellulaires, pseudohyphales, voire d’hyphe, et
d’une matrice extracellulaire composée de
polysaccharides et de protéines. Ce biofilm protège
notamment C. albicans contre les antifongiques. La formation
du biofilm s’effectue en trois étapes : des colonies
unicellulaires commencent par se former; ensuite les cellules
adoptent une conformation pseudohyphale et produisent une matrice
extracellulaire. Finalement, matrice et filaments (hyphe) grandissent
de concert.
Son génome représente 16 Mb (haploïde) pour
~6500 gènes (dont 200 env. contiennent des introns ; taille
moyenne des ORF : ~1.5kb ; lien)
répartis sur 8 paires de chromosomes homologues ; il a été
entièrement séquencé en 1998 (lien
; lien). Les
analyses génétiques de Candida albicans sont
difficiles car ce champignon grandit sous forme diploïde asexuée
(Pourquoi cela pose-t-il un problème
?).
|
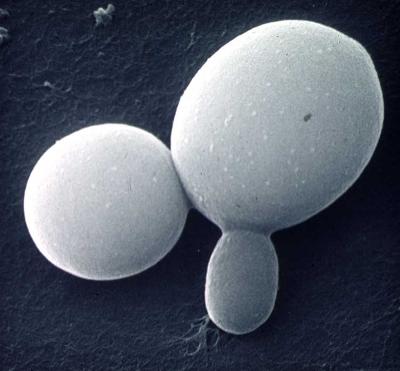
Forme planctonique
|
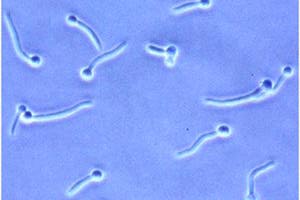
Forme pseudohyphale
|

Forme hyphale
|
Plus d’info sur Candida albicans :
http://www.pasteur.fr/recherche/unites/RIF//lie.html
Plus d’info sur les candidoses :
http://www.cnrs.fr/SDV/Dept/candidose.pdf
;
http://www.med.univ-rennes1.fr/cgi-bin/iw/fichiers_de_codeadm.pl?code=M01219
Banque génomique :
Une banque génomique est une collection de plasmides, dont
chacun contient un morceau de chromosome provenant de l’organisme
que l’on souhaite étudier. Une banque génomique
idéale couvre 10 fois le génome complet, avec des
fragments « chevauchant » et de différentes
longueurs. Une banque génomique peut servir de support pour
séquencer le génome de cet organisme, de banque de
complémentation afin d’identifier des mutants récessifs
(p.ex dans l’expérience
du TD2), ou pour la création d’une banque
d’insertion (inactivation de gène par insertion d’un
marqueur).
Questions : Pourquoi est-il important
de couvrir plus d’une fois le génome ? Quel
est l’intérêt d’avoir des fragments
chevauchants ?
Banque d’insertion :
Il y a plusieurs façons de créer une banque
d’insertion, mais cela n’étant pas le sujet de ce
TD, nous ne verrons que celle utilisée dans les articles
cités (et sur lesquels se base cette petite expérience).
A partir d’une banque génomique, des insertions par un
transposon (qui inclut un marqueur de sélection) sont
réalisées in vitro dans les fragments
chromosomiques de la banque. Après amplification de la banque
(par des bactéries), les fragments seront excisés des
vecteurs et utilisés pour transformer des cellules de Candida
albicans (création d’une collection de cellules
mutantes par insertion ; par recombinaison, les insertions
remplaceront les copies chromosomiques). Chaque transformant sera
alors étudié pour découvrir le phénotype
recherché. Le locus de l’insertion sera alors identifié
par séquençage.
Question : Voyez vous d’autres
moyens de procéder pour fabriquer une collection de mutations
par insertion ?
Expérience 1
Première étape : Préparation du
vecteur.
Durant cette étape, vous allez choisir un vecteur et une
enzyme de restriction pour couper votre vecteur (qui recevra les
fragments de chromosomes de Candida albicans).
Pour construire votre banque deux vecteurs s’offrent à
vous : (cliquez sur la bonne image)
|
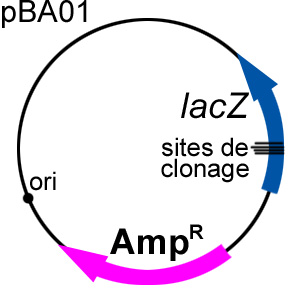
pBA01 (nom fictif), un plasmide (dérivé
de pUC19) disposant d’une origine de réplication
bactérienne «high copy» (dans une
bactérie, il y aura entre 75 et >200 copies du vecteur
selon la température), d’un gène bactérien
de résistance à un antibiotique et, d’un site
de clonage (contenant plusieurs sites de restriction) inséré
dans l’ORF du gène rapporteur lacZ.
|
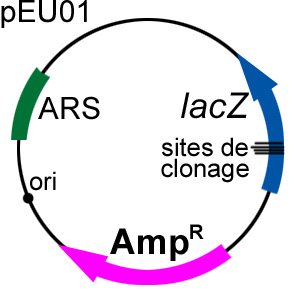
pEU01 (nom fictif), un plasmide disposant
d’une origine de réplication bactérienne «low
copy» (dans une bactérie, il y aura entre 5 et
10 copies du vecteur), d’un gène bactérien de
résistance à un antibiotique, mais également
d’une origine de réplication eucaryotique (de S.
cerevisiae, mais qui fonctionne également chez C.
albicans) et, d’un site de clonage (contenant plusieurs
sites de restriction) inséré dans l’ORF du
gène rapporteur lacZ.
|
En vous aidant des informations données sur la construction
de la banque d’insertion dans la partie Introduction
et des quelques questions qui suivent, choisissez le vecteur que vous
allez utiliser pour construire votre banque génomique.
Questions à se poser : Allez-vous transformer votre
vecteur dans les bactéries ET/OU Candida albicans. Avez vous
besoin d’une bonne amplification par les bactéries de
votre vecteur (high copy) ou non (low copy).
B. Emery. 5.11.08 - mise à jour des liens